Журнал «Здоровье ребенка» Том 16, №3, 2021
Вернуться к номеру
Біогенез мікроРНК. Частина 2. Формування зрілих мікроРНК. Матурація неканонічних мікроРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді наведений біогенез мікроРНК. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. У статті відображені етапи формування зрілих мікроРНК. Зазначено, що утворені в результаті DICER-опосередкованого розщеплення дуплексні РНК взаємодіють із протеїнами AGO, формуючи ефекторний РНК-індукований сайленсинговий комплекс (RISC). Зазначено, що дефіцит протеїнів AGO призводить до помітного зменшення кількості мікроРНК, а надлишкова експресія білків AGO супроводжується підвищенням рівня мікроРНК. Наведені основні етапи збірки повністю функціонального RISC. Перший етап — завантаження дуплексних мікроРНК на протеїни AGO. Другий етап — розкручування дуплексних мікроРНК. Наведені захворювання людини, що асоційовані з порушенням процесингу в цитоплазмі клітини. Дана характеристика численним альтернативним механізмам, які задіяні у формуванні функціонально активних мікроРНК. Розрізняють три класи міртронів: типові, 5’-хвостові і 3’-хвостові. Ендогенні кшРНК нагадують Drosha-незалежні синтетичні кшРНК, що використані для експериментальної індукції нокауту генів. Химерні шпильки неканонічних генів мікроРНК транскрибуються в тандемі або як частина іншого типу гена малої РНК. Таким чином, формування зрілих мікроРНК відбувається за рахунок утворення комплексу RISC. Ядро комплексу RISC складається з мікроРНК, AGO і протеїну з тринуклеотидним повтором 6. Завантаження дцРНК на протеїни AGO і подальше розкручування дуплексних РНК становлять основні етапи збірки повністю функціонального RISC. Порушення процесингу в цитоплазмі клітини асоційоване з розвитком деяких захворювань людини. Існують альтернативні механізми, які задіяні у формуванні функціонально активних мікроРНК: міртронів, ендогенних коротких РНК, що містять шпильки, химерних шпильок.
The scientific review presents the biogenesis of miRNAs. To write the article, information was searched using databases Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. The article shows the stages of formation of mature miRNAs. It is noted that duplex RNAs resulting from DICER-mediated cleavage interact with Argonaute (AGO) proteins to form an effector RNA-induced silencing complex (RISC). It is shown that the deficiency of AGO proteins leads to a significant decrease in the amount of miRs, and overexpression of AGO proteins is accompanied by an increase in the level of miRs. The main stages of assembling a fully functional RISC are presented. The first stage is the loading of duplex miRs on AGO proteins. The second stage is the promotion of duplex miRs. Human diseases associated with processing disorders in the cytoplasm of the cell are presented. Numerous alternative mechanisms involved in the formation of functionally active miRs are is characterized. There are three classes of mirtrons: typical mirtrons, 5’-tailed mirtrons and 3’-tailed mirtrons. Endogenous csRNAs resemble Drosha-independent synthetic csRNAs used to experimentally induce gene knockout. Chimeric hairpins of non-canonical miR genes are transcribed in tandem or as a part of another type of small RNA gene. Thus, the formation of mature miRs occurs due to the formation of the RISC complex. The core of the RISC complex consists of microRNA, AGO and protein with a trinucleotide repeat 6. Loading dsRNA on AGO proteins and subsequent promotion of duplex RNA are the main stages of assembly of a fully functional RISC. Disorders of processing in the cytoplasm of the cell are associated with the development of some human diseases. There are alternative mechanisms involved in the formation of functionally active miRs: mirtrons, endogenous short RNAs containing hairpins, chimeric hairpins.
мікроРНК; матурація мікроРНК; протеїни Argonaute (AGO); РНК-індукований сайленсинговий комплекс (RISC); огляд
microRNA (miRNA; miR); maturation of miRNA; Argonaute proteins; RNA-induced silencing complex; review
Формування зрілих мікроРНК
/62.jpg)
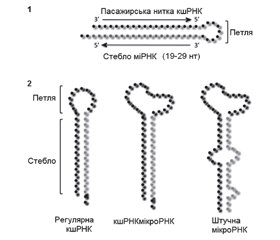
Матурація неканонічних мікроРНК (альтернативні шляхи матурації мікроРНК)
/63.jpg)
/65.jpg)
Висновки
- Azlan A., Dzaki N., Azzam G. Argonaute: The executor of small RNA function. J. Genet. Genomics. 2016, Aug 20. 43(8). 481-494. doi: 10.1016/j.jgg.2016.06.002.
- Bartel D.P. Metazoan MicroRNAs. Cell. 2018, Mar 22. 173(1). 20-51. doi: 10.1016/j.cell.2018.03.006.
- Bianchi N., Zuccato C., Finotti A. et al. Involvement of miRNA in erythroid differentiation. Epigenomics. 2012 Feb. 4(1), 51-65. doi: 10.2217/epi.11.104.
- Caimari F., Kumar A.V., Kurzawinski T. et al. A novel DICER 1 mutation in familial multinodular goiter. Clin. Endocrinol. (Oxf.) 2018, Apr 7. doi: 10.1111/cen.13613.
- Caviglia J.M., Yan J., Jang M.K. et al. MicroRNA-21 and Dicer are dispensable for hepatic stellate cell activation and the development of liver fibrosis. Hepatology. 2017, Nov 1. doi: 10.1002/hep.29627.
- Cheloufi S., Dos Santos C.O., Chong M.M., Hannon G.J. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis. Nature. 2010, Jun 3. 465(7298). 584-589. doi: 10.1038/nature09092.
- Connerty P., Ahadi A., Hutvagner G. RNA Binding Proteins in the miRNA Pathway. Int. J. Mol. Sci. 2015, Dec 26. 17(1). pii: E31. doi: 10.3390/ijms17010031.
- Cullen B.R., Cherry S., tenOever B.R. Is RNA interference a physiologically relevant innate antiviral immune response in mammals? Cell Host Microbe. 2013, Oct 16. 14(4). 374-378. doi: 10.1016/j.chom.2013.09.011.
- Curtis H.J., Sibley C.R., Wood M.J. Mirtrons, an emerging class of atypical miRNA. Wiley Interdiscip Rev RNA. 2012 Sep-Oct. 3(5). 617-632. doi: 10.1002/wrna.1122.
- Dueck A., Ziegler C., Eichner A. et al. microRNAs associated with the different human Argonaute proteins. Nucleic Acids Res. 2012 Oct. 40(19). 9850-9862. doi: 10.1093/nar/gks705.
- Foulkes W.D., Priest J.R., Duchaine T.F. DICER1: mutations, microRNAs and mechanisms. Nat. Rev. Cancer. 2014 Oct. 14(10). 662-672. doi: 10.1038/nrc3802.
- Gomez-Escobar N., Almobadel N., Alzahrani O. et al. Translin and Trax differentially regulate telomere-associated transcript homeostasis. Oncotarget. 2016, Jun 7. 7(23). 33809-33820. doi: 10.18632/oncotarget.9278.
- Ha M., Kim V.N. Regulation of microRNA biogenesis. Nat. Rev. Mol. Cell Biol. 2014 Aug. 15(8). 509-524. doi: 10.1038/nrm3838.
- Hata A., Kashima R. Dysregulation of microRNA biogenesis machinery in cancer. Crit. Rev. Biochem. Mol. Biol. 2016 May-Jun. 51(3). 121-134. doi: 10.3109/10409238.2015.1117054.
- Herrera-Carrillo E., Berkhout B. Dicer-independent processing of small RNA duplexes: mechanistic insights and applications. Nucleic Acids Res. 2017, Oct 13. 45(18). 10369-10379. doi: 10.1093/nar/gkx779.
- Janas M.M., Wang B., Harris A.S. et al. Alternative RISC assembly: binding and repression of microRNA-mRNA duplexes by human Ago proteins. RNA. 2012 Nov. 18(11). 2041-2055. doi: 10.1261/rna.035675.112.
- Juvvuna P.K., Khandelia P., Lee L.M., Makeyev E.V. Argonaute identity defines the length of mature mammalian microRNAs. Nucleic Acids Res. 2012 Aug. 40(14). 6808-6820. doi: 10.1093/nar/gks293.
- Kawamata T., Tomari Y. Making RISC. Trends Biochem. Sci. 2010 Jul. 35(7). 368-376. doi: 10.1016/j.tibs.2010.03.009.
- Kim H., Kim J., Yu Sh., Lee Y-Y., Park J. at al. A Mechanism for microRNA Arm Switching Regulated by Uridylation. Molecular Сell. 2020. 6(78). 1224-1236. https://doi.org/10.1016/j.molcel.2020.04.030.
- Kim J.O., Bae J., Kim J. et al. Association of MicroRNA Biogenesis Genes Polymorphisms with Ischemic Stroke Susceptibility and Post-Stroke Mortality. J. Stroke. 2018 Jan. 20(1). 110-121. doi: 10.5853/jos.2017.02586.
- King V.M., Borchert G.M. MicroRNA Expression: Protein Participants in MicroRNA Regulation. Methods Mol. Biol. 2017. 1617. 27-37. doi: 10.1007/978-1-4939-7046-9_2.
- Klein M., Chandradoss S.D., Depken M., Joo C. Why Argonaute is needed to make microRNA target search fast and reliable. Semin. Cell Dev. Biol. 2017 May. 65. 20-28. doi: 10.1016/j.semcdb.2016.05.017.
- Kobayashi H., Tomari Y. RISC assembly: Coordination between small RNAs and Argonaute proteins. Biochim. Biophys. Acta. 2016 Jan. 1859(1). 71-81. doi: 10.1016/j.bbagrm.2015.08.007.
- Ladewig E., Okamura K., Flynt A.S. et al. Discovery of hundreds of mirtrons in mouse and human small RNA data. Genome Res. 2012 Sep. 22(9). 1634-1645. doi: 10.1101/gr.133553.111.
- Lambeth L.S., Smith C.A. Short hairpin RNA-mediated gene silencing. Methods Mol. Biol. 2013. 942. 205-232. doi: 10.1007/978-1-62703-119-6_12.
- Langenberger D., Çakir M.V., Hoffmann S., Stadler P.F. Dicer-processed small RNAs: rules and exceptions. J. Exp. Zool. B Mol. Dev. Evol. 2013 Jan. 320(1). 35-46. doi: 10.1002/jez.b.22481.
- Medley J.C., Panzade G., Zinovyeva A.Y. microRNA strand selection: Unwinding the rule. WIREs RNA. 2020. https://doi.org/10.1002/wrna.1627.
- Nakanishi K. Anatomy of RISC: how do small RNAs and chaperones activate Argonaute proteins? Wiley Interdiscip Rev. RNA. 2016 Sep. 7(5). 637-660. doi: 10.1002/wrna.1356.
- Okamura K., Hagen J.W., Duan H. et al. The mirtron pathway generates microRNA-class regulatory RNAs in Drosophila. Cell. 2007, Jul 13. 130(1). 89-100. doi: 10.1016/j.cell.2007.06.028.
- Sheu-Gruttadauria J., MacRae I.J. Structural Foundations of RNA Silencing by Argonaute. J. Mol. Biol. 2017, Aug 18. 429(17). 2619-2639. doi: 10.1016/j.jmb.2017.07.018.
- Tarallo V., Hirano Y., Gelfand B.D. et al. DICER1 loss and Alu RNA induce age-related macular degeneration via the NLRP3 inflammasome and MyD88. Cell. 2012, May 11. 149(4). 847-859. doi: 10.1016/j.cell.2012.03.036.
- Titov I.I., Vorozheykin P.S. Comparing miRNA structure of mirtrons and non-mirtrons. BMC Genomics. 2018, Feb 9. 19(Suppl. 3). 114. doi: 10.1186/s12864-018-4473-8.
- Tokiyoshi E., Watanabe M., Inoue N. et al. Polymorphisms and expression of genes encoding Argonautes 1 and 2 in autoimmune thyroid diseases. Autoimmunity. 2018 Feb. 51(1). 35-42. doi: 10.1080/08916934.2017.1416468.
- Westholm J.O., Lai E.C. Mirtrons: microRNA biogenesis via splicing. Biochimie. 2011 Nov. 93(11). 1897-1904. doi: 10.1016/j.biochi.2011.06.017.
- Yang J.S., Lai E.C. Alternative miRNA biogenesis pathways and the interpretation of core miRNA pathway mutants. Mol. Cell. 2011, Sep 16. 43(6). 892-903. doi: 10.1016/j.molcel.2011.07.024.

/61.jpg)
/62_2.jpg)
/63_2.jpg)
/64.jpg)