Журнал «Здоровье ребенка» Том 16, №4, 2021
Вернуться к номеру
Регуляція вмісту мікроРНК. Частина 1. Редагування мікроРНК. Тайлінг мікроРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді наведено процеси регуляції вмісту мікроРНК. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. У статті наведена характеристика процесів редагування мікроРНК та тайлінгу мікроРНК, що регулюють вміст мікроРНК за рахунок контролю над транскрипцією. Підкреслено, що редагування є найважливішим механізмом посттранскрипційної регуляції мікроРНК та відбувається за допомогою РНК-специфічної аденозиндезамінази. У статті відображено, що редагування призводить до зміни вторинної структури молекули мікроРНК і девіації процесу дозрівання мікроРНК. Наведено, що тайлінг мікроРНК являє собою посттранскрипційне подовження хвоста молекули за рахунок додавання нуклеотидів до 3’-кінця РНК. Розкрито, що уридинілірування є значно поширеним посттранскрипційним процесом, що регулює експресію генів. На прикладі родини miR let-7, які пригнічують проліферацію і сприяють диференціюванню клітин, продемонстровано вплив уридинілірування на біогенез мікроРНК. Установлено, що інший тип тайлінгу мікроРНК, а саме аденілірування найчастіше сприяє стабілізації молекули, але в деяких випадках може призвести і до деградації мікроРНК. Таким чином, регуляція вмісту мікроРНК здійснюється за допомогою редагування мікроРНК, тайлінгу мікроРНК. За рахунок редагування відбуваються зміна вторинної структури молекули мікроРНК і девіація процесу дозрівання мікроРНК. Тайлінг мікроРНК являє собою посттранскрипційне подовження хвоста молекули за рахунок додавання нуклеотидів до 3’-кінця РНК за допомогою поліуридинілірування або поліаденірування. Уридинілірування впливає на процесинг і деградацію попередників мікроРНК із різними молекулярними наслідками, які в деяких випадках сприяють розвитку захворювань.
This scientific review presents the processes of regulation of miRNA content. To write the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. The article presents the characteristics of the processes of microRNA editing and microRNA tailing, which regulate the content of microRNA through the transcription control. It is emphasized that editing is the most important mechanism of posttranscriptional regulation of microRNA that occurs with the help of RNA-specific adenosine deaminase. The article shows that editing leads to a change in the secondary structure of the microRNA molecule and the deviation of the process of microRNA maturation. It is shown that microRNA tailing is a posttranscriptional elongation of the tail of the molecule by adding nucleotides to the 3’-end of RNA. It has been found that uridinylation is a very common posttranscriptional process that regulates gene expression. The effect of uridinylation on microRNA biogenesis has been demonstrated on the example of the miR let-7 family, which inhibits proliferation and promotes cell differentiation. It has been established that another type of microRNA tailing, namely adenylation, most often contributes to the stabilization of the molecule, but in some cases can lead to microRNA degradation. Thus, the regulation of miRNA content is carried out by editing miRNA, microRNA tailing. Due to editing, the secondary structure of the microRNA molecule changes and the microRNA maturation process deviates. MicroRNA tailing is a post-transcriptional elongation of the tail of the molecule by adding nucleotides to the 3’-end of RNA by polyuridinylation or polyadenylation. Uridinylation affects the processing and degradation of miRNA precursors with different molecular effects, which in some cases contribute to the development of diseases.
мікроРНК; редагування мікроРНК; тайлінг мікроРНК; огляд
microRNA (miRNA, miR); еditing miRNA; tailing miRNA; review
Вступ
Редагування мікроРНК
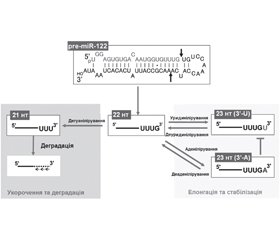
Тайлінг мікроРНК
Висновок
- Bahn J.H., Ahn J., Lin X. et al. Genomic analysis of ADAR1 binding and its involvement in multiple RNA processing pathways. Nat. Commun. 2015, Mar 9. 6. 6355. doi: 10.1038/ncomms7355.
- Boele J., Persson H., Shin J.W. et al. PAPD5-mediated 3' adenylation and subsequent degradation of miR-21 is disrupted in proliferative disease. Proc. Natl Acad. Sci. USA. 2014, Aug 5. 111(31). 11467-11472. doi: 10.1073/pnas.1317751111.
- Burroughs A.M. Ando Y., de Hoon M.J. et al. A comprehensive survey of 3' animal miRNA modification events and a possible role for 3' adenylation in modulating miRNA targeting effectiveness. Genome Res. 2010 Oct. 20(10). 1398-1410. doi: 10.1101/gr.106054.110.
- Cai Y., Yu X., Hu S., Yu J. A brief review on the mechanisms of miRNA regulation. Genomics Proteomics Bioinformatics. 2009 Dec. 7(4). 147-154. doi: 10.1016/S1672-0229(08)60044-3.
- Chung C.Z., Seidl L.E., Mann M.R., Heinemann I.U. Tipping the balance of RNA stability by 3' editing of the transcriptome. Biochim. Biophys. Acta. 2017 Nov. 1861(11 Pt B). 2971-2979. doi: 10.1016/j.bbagen.2017.05.003.
- Correia M. de Sousa, Gjorgjieva M., Dolicka D., Sobolewski C., Foti M. Deciphering miRNAs’ Action through miRNA Editing. International Journal of Molecular Sciences. 2019. 20(24). 6249. https://doi.org/10.3390/ijms20246249.
- De Almeida C., Scheer Н., Zuber Н., Gagliardi D. RNA uridylation: a key posttranscriptional modification shaping the coding and noncoding transcriptome. Wiley Interdiscip. Rev. RNA. 2018 Jan. 9(1). doi: 10.1002/wrna.1440.
- Esau C., Davis S., Murray S.F. et al. miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting. Cell. Metab. 2006 Feb. 3(2). 87-98. doi: 10.1016/j.cmet.2006.01.005.
- Frederick M., Heinemann I. Regulation of RNA stability at the 3′ end// Biological Chemistry. 2021. 402(4). 425-431. https://doi.org/10.1515/hsz-2020-0325.
- Haldipur B., Bhukya P.L., Arankalle V., Lole K. Positive Regulation of Hepatitis E Virus Replication by MicroRNA-122. J. Virol. 2018, May 14. 92(11). pii: e01999-17. doi: 10.1128/JVI.01999-17.
- Katoh T., Sakaguchi Y., Miyauchi K. et al. Selective stabilization of mammalian microRNAs by 3' adenylation mediated by the cytoplasmic poly(A) polymerase GLD-2. Genes. Dev. 2009, Feb 15. 23(4). 433-438. doi: 10.1101/gad.1761509.
- Katoh T., Hojo H., Suzuki T. Destabilization of microRNAs in human cells by 3' deadenylation mediated by PARN and CUGBP1. Nucleic Acids Res. 2015, Sep 3. 43(15). 7521-7534. doi: 10.1093/nar/gkv669.
- Kim H., Kim J., Yu Sh., Lee Y-Y., Park J. at al. A Mechanism for microRNA Arm Switching Regulated by Uridylation. Molecular Сell. 2020. 6(78). 1224-1236. https://doi.org/10.1016/j.molcel.2020.04.030.
- King V.M., Borchert G.M. MicroRNA Expression: Protein Participants in MicroRNA Regulation. Methods Mol. Biol. 2017. 1617. 27-37. doi: 10.1007/978-1-4939-7046-9_2.
- Koyano K., Bahn J.H., Xiao X. Extracellular microRNA 3’ end modification across diverse body fluids. Epigenetics. 2020. doi: 10.1080/15592294.2020.1834922.
- Lee H., Han S., Kwon C.S., Lee D. Biogenesis and regulation of the let-7 miRNAs and their functional implications. Protein Cell. 2016 Feb. 7(2). 100-113. doi: 10.1007/s13238-015-0212-y.
- Lehmann K.A., Bass B.L. Double-stranded RNA adenosine deaminases ADAR1 and ADAR2 have overlapping specificities. Biochemistry. 2000, Oct 24. 39(42). 12875-12884. PMID: 11041852.
- Liu W.H., Chen C.H., Yeh K.H. et al. ADAR2-mediated editing of miR-214 and miR-122 precursor and antisense RNA transcripts in liver cancers. PLoS One. 2013, Dec 27. 8(12). e81922. doi: 10.1371/journal.pone.0081922.
- Marceca G.P., Distefano R., Tomasello L., Lagana A., Russo F., Calore F., Romano G. еt al. MiREDiBase: a manually curated database of validated and putative editing events in microRNAs. bioRxiv. doi: https://doi.org/10.1101/2020.09.04.283689.
- Munoz-Tello P., Rajappa L., Coquille S., Thore S. Polyuridylation in Eukaryotes: A 3'-End Modification Regulating RNA Life. Biomed. Res. Int. 2015. 2015. 968127. doi: 10.1155/2015/968127.
- Nakano M., Nakajima M. Significance of A-to-I RNA editing of transcripts modulating pharmacokinetics and pharmacodynamics. Pharmacol. Ther. 2018 Jan. 181. 13-21. doi: 10.1016/j.pharmthera.2017.07.003.
- Niepmann M., Shalamova L.A., Gerresheim G.K., Rossbach O. Signals Involved in Regulation of Hepatitis C Virus RNA Genome Translation and Replication. Front. Microbiol. 2018, Mar 12. 9. 395. doi: 10.3389/fmicb.2018.00395.
- Nishikura K. A-to-I editing of coding and non-coding RNAs by ADARs. Nat. Rev. Mol. Cell. Biol. 2016 Feb. 17(2). 83-96. doi: 10.1038/nrm.2015.4.
- Ota H., Sakurai M., Gupta R. et al. ADAR1 forms a complex with Dicer to promote microRNA processing and RNA-induced gene silencing. Cell. 2013, Apr 25. 153(3). 575-589. doi: 10.1016/j.cell.2013.03.024.
- Pashler A.L., Towler B.P., Jones C.I., Newbury S.F. The roles of the exoribonucleases DIS3L2 and XRN1 in human disease. Biochem. Soc. Trans. 2016, Oct 15. 44(5). 1377-1384.
- Pinto Y., Buchumenski I., Levanon E.Y., Eisenberg E. Human cancer tissues exhibit reduced A-to-I editing of miRNAs coupled with elevated editing of their targets. Nucleic Acids Res. 2018, Jan 9. 46(1). 71-82. doi: 10.1093/nar/gkx1176.
- Scheer H., Zuber H., De Almeida C., Gagliardi D. Uridylation Earmarks mRNAs for Degradation… and More. Trends Genet. 2016 Oct. 32(10). 607-619. doi: 10.1016/j.tig.2016.08.003.
- Slezak-Prochazka I., Durmus S., Kroesen B.J., van den Berg A. MicroRNAs, macrocontrol: regulation of miRNA processing. RNA. 2010 Jun. 16(6). 1087-1095. doi: 10.1261/rna.1804410.
- Song J., Song J., Mo B., Chen X. Uridylation and adenylation of RNAs. Sci. China Life Sci. 2015 Nov. 58(11). 1057-1066. doi: 10.1007/s11427-015-4954-9.
- Tan M.H., Li Q., Shanmugam R. et al. Dynamic landscape and regulation of RNA editing in mammals. Nature. 2017, Oct 11. 550(7675). 249-254. doi: 10.1038/nature24041.
- Towler B.P., Jones C.I., Viegas S.C. et al. The 3’-5’ exoribonuclease Dis3 regulates the expression of specific microRNAs in Drosophila wing imaginal discs. RNA Biol. 2015. 12(7). 728-741. doi: 10.1080/15476286.2015.1040978.
- Wang Y., Liang H. When MicroRNAs Meet RNA Editing in Cancer: A Nucleotide Change Can Make a Difference. Bioessays. 2018 Feb. 40(2). doi: 10.1002/bies.201700188.
- Yamada H., Ohashi K., Suzuki K. et al. Longitudinal study of circulating miR-122 in a rat model of non-alcoholic fatty liver disease. Clin. Chim. Acta. 2015, Jun 15. 446. 267-271. doi: 10.1016/j.cca.2015.05.002.
- Yu S., Kim V.N. A tale of non-canonical tails: gene regulation by post-transcriptional RNA tailing. Nat. Rev. Mol. Cell. Biol. 2020. 21. 542-556. https://doi.org/10.1038/s41580-020-0246-8.
- Zhang J., Wang Q., Zhao X. et al. MicroRNA-122 targets genes related to goose fatty liver. Poult. Sci. 2018, Feb 1. 97(2). 643-649. doi: 10.3382/ps/pex307.
- Zhao S., Liu M.F. Mechanisms of microRNA-mediated gene regulation. Sci. China Life Sci. 2009 Dec. 52(12). 1111-1116. doi: 10.1007/s11427-009-0152-y.

/81.jpg)
/82.jpg)
/82_2.jpg)
/83_2.jpg)
/83.jpg)
/84.jpg)
/85.jpg)