Журнал «Здоровье ребенка» Том 16, №7, 2021
Вернуться к номеру
Локалізація та транслокація зрілих мікроРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
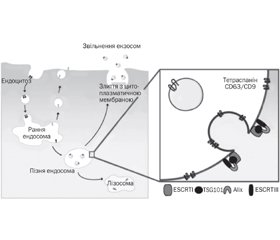
У науковому огляді наведені шляхи ядерного імпорту та експорту мікроРНК у клітині. Авторами надана чітка та доступна схема транслокації мікроРНК у клітині. У статті відображено, що основним місцем локалізації в цитоплазмі клітини комплексу RISC і його компонентів, у тому числі і мікроРНК, є процесингові P-тільця. Автори наводять факт того, що і протеїни Argonaute — сигнатурні компоненти ефекторного комплексу інтерференції РНК RISC локалізуються в P-тільцях ссавців. Відомо, що протеїни родини каріоферинів опосередковують транслокацію miRISC в ядро клітини. Дані протеїни розпізнають послідовності ядерної локалізації в амінокислотних послідовностях протеїнів й активно транспортують дані протеїни через пори ядерної мембрани клітини. Підкреслено, що, крім неселективних механізмів ядерного імпорту мікроРНК, існують і транспортні механізми, які переносять певні мікроРНК через мембрану клітини. Наведені деякі мікроРНК, що переважно локалізуються в ядрі певного типу клітин. Науковці вважають, що значна частина мікроРНК ядра сконцентрована в полісомах. Експорт мікроРНК ядерного пулу в цитоплазму клітини відбувається за допомогою експортину-1. Таким чином, в цитоплазмі клітини накопичуються зрілі форми мікроРНК, частина з яких транслокується в ядро клітини або в екстрацелюлярний простір. Складання комплексу miRISC здійснюється в цитоплазмі клітини, і тільки після формування комплексу відбувається його імпорт в ядро клітини. Спектр екзосомасоційованих мікроРНК може бути високозначущим діагностичним критерієм для деяких нозологій, а екзосоми, що містять певні мікроРНК, можуть використовуватися для цілеспрямованої терапії конкретних захворювань. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka.
The scientific review shows the ways of nuclear import and export of miRNAs in the cell. The authors present a clear and accessible scheme of microRNA translocation in the cell. The article shows that the main site of localization in the cytoplasm of cells of the RISC complex and its components, including miRNAs, are processing P-cells. The authors cite the fact that Argonaute proteins — signature components of the effector complex of RISC RNA interference — are localized in mammalian P-bodies. It is shown that proteins of the karyopherin family mediate the translocation of miRISC into the cell nucleus. These proteins recognize nuclear localization sequences (NLS) in the amino acid sequences of proteins and actively transport these proteins through the pores of the cell’s nuclear membrane. It is emphasized that in addition to non-selective mechanisms of nuclear import of miRNAs, there are transport mechanisms that carry certain miRNAs across the cell membrane. Some miRNAs are presented, which are mainly localized in the nucleus of a certain type of cell. Scientists believe that much of the nucleus miRNA is concentrated in polysomes. Export of nuclear pool microRNA into the cytoplasm of the cell occurs with the help of export 1. Thus, in the cytoplasm of the cell, mature forms of microRNA accumulate, some of which are translocated to the cell nucleus or the extracellular space. Assembly of the miRISC complex is carried out in the cytoplasm of the cell, and only after the formation of the complex, it is imported into the cell nucleus. The spectrum of exosome-associated miRNAs can be a highly important diagnostic criterion for some nosologies, and exosomes containing certain miRNAs can be used for targeted therapy of specific diseases. To write the article, information was searched using databases Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka.
мікроРНК; транслокація мікроРНК; P-тільця; локалізація мікроРНК; РНК-індукований сайленсинговий комплекс; огляд
microRNA; microRNA translocation; P-bodies; microRNA localization; RNA-induced silencing complex (RISC); review
Вступ
Цитоплазматична локалізація зрілих мікроРНК
Імпорт мікроРНК в ядро клітини
Ядерна локалізація мікроРНК
Експорт мікроРНК з ядра клітини
Екстрацелюлярні мікроРНК
Висновки
- Akers J.C., Gonda D., Kim R. et al. Biogenesis of extracellular vesicles (EV): exosomes, microvesicles, retrovirus-like vesicles, and apoptotic bodies. J. Neurooncol. 2013 May. 113(1). 1-11. doi: 10.1007/s11060-013-1084-8.
- Arroyo J.D., Chevillet J.R., Kroh E.M. et al. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc. Natl. Acad. Sci. USA. 2011. 108. 5003-5008. doi: 10.1073/pnas.1019055108.
- Barrès C., Blanc L., Bette-Bobillo P. et al. Galectin-5 is bound onto the surface of rat reticulocyte exosomes and modulates vesicle uptake by macrophages. Blood. 2010, Jan 21. 115(3). 696-705. doi: 10.1182/blood-2009-07-231449.
- Bayraktar R., Van Roosbroeck K., Calin G.A. Cell-to-cell communication: microRNAs as hormones. Mol. Oncol. 2017 Dec. 11(12). 1673-1686. doi: 10.1002/1878-0261.12144.
- Bhatnagar S., Schorey J.S. Exosomes released from infected macrophages contain Mycobacterium avium glycopeptidolipids and are proinflammatory. J. Biol. Chem. 2007, Aug 31. 282(35). 25779-89. doi: 10.1074/jbc.M702277200.
- Bhome R., Del Vecchio F., Lee G.H. et al. Exosomal microRNAs (exomiRs): Small molecules with a big role in cancer. Cancer Lett. 2018, Apr 28. 420. 228-235. doi: 10.1016/j.canlet.2018.02.002.
- Blandford S.N., Galloway D.A., Moore C.S. The roles of extracellular vesicle microRNAs in the central nervous system. Glia. 2018, May 4. doi: 10.1002/glia.23445.
- Bolukbasi M.F., Mizrak A., Ozdener G.B. et al. miR-1289 and "Zipcode"-like Sequence Enrich mRNAs in Microvesicles. Mol. Ther. Nucleic. Acids. 2012, Feb 7. 1. e10. doi: 10.1038/mtna.2011.2.
- Carretero-González A., Otero I., Carril-Ajuria L. et al. Exosomes: Definition, Role in Tumor Development and Clinical Implications. Cancer Microenviron. 2018, May 3. doi: 10.1007/s12307-018-0211-7.
- Chen L., Chen R., Kemper S. et al. Therapeutic effects of serum extracellular vesicles in liver fibrosis. J. Extracell. Vesicles. 2018, Apr 17. 7(1). 1461505. doi: 10.1080/20013078.2018.1461505.
- da Silveira J.C., de Ávila A.C.F.C.M., Garrett H.L. et al. Cell-secreted vesicles containing microRNAs as regulators of gamete maturation. J. Endocrinol. 2018 Jan. 236(1). R15-R27. doi: 10.1530/JOE-17-0200.
- de Jong O.G., Verhaar M.C., Chen Y.et al Cellular stress conditions are reflected in the protein and RNA content of endothelial cell-derived exosomes. J. Extracell. Vesicles. 2012, Apr 16. 1. doi: 10.3402/jev.v1i0.18396.
- Deng H., Sun C., Sun Y. et al. Lipid, Protein, and MicroRNA Composition Within Mesenchymal Stem Cell-Derived Exosomes. Cell. Reprogram. 2018 Jun. 20(3). 178-186. doi: 10.1089/cell.2017.0047.
- Devhare P.B., Ray R.B. Extracellular vesicles: Novel mediator for cell to cell communications in liver pathogenesis. Mol. Aspects Med. 2018 Apr. 60. 115-122. doi: 10.1016/j.mam.2017.11.001.
- Gagnon K.T., Li L., Chu Y. et al. RNAi factors are present and active in human cell nuclei. Cell. Rep. 2014, Jan 16. 6(1). 211-21. doi: 10.1016/j.celrep.2013.12.013.
- Gallagher C., Ramos A. Joining the dots — protein-RNA interactions mediating local mRNA translation in neurons. FEBS Lett. 2018, Jun 1. doi: 10.1002/1873-3468.13121.
- Gibbings D.J., Ciaudo C., Erhardt M., Voinnet O. Multivesicular bodies associate with components of miRNA effector complexes and modulate miRNA activity. Nat. Cell. Biol. 2009 Sep. 11(9). 1143-9. doi: 10.1038/ncb1929.
- Groot M., Lee H. Sorting Mechanisms for MicroRNAs into Extracellular Vesicles and Their Associated Diseases. Cells. 2020. 9(4). 1044. https://doi.org/10.3390/cells9041044.
- Hao S., Bai O., Li F. et al. Mature dendritic cells pulsed with exosomes stimulate efficient cytotoxic T-lymphocyte responses and antitumour immunity. Immunology. 2007 Jan. 120(1). 90-102. doi: 10.1111/j.1365-2567.2006.02483.x.
- Heijnen H., van der Sluijs P. Platelet secretory behaviour: as diverse as the granules… or not? J. Thromb. Haemost. 2015 Dec. 13(12). 2141-51. doi: 10.1111/jth.13147.
- Hess C., Sadallah S., Hefti A. et al. Ectosomes released by human neutrophils are specialized functional units. J. Immunol. 1999, Oct 15. 163(8). 4564-73.
- Hwang H.W., Wentzel E.A., Mendell J.T. A hexanucleotide element directs microRNA nuclear import. Science. 2007, Jan 5. 315(5808). 97-100. Doi: 10.1126/science.1136235.
- Johnstone R.M., Adam M., Hammond J.R. et al. Vesicle formation during reticulocyte maturation. Association of plasma membrane activities with released vesicles (exosomes). J. Biol. Chem. 1987, Jul 5. 262(19). 9412-20. PMID: 3597417.
- Kamenska A., Simpson C., Vindry C. et al. The DDX6-4E-T interaction mediates translational repression and P-body assembly. Nucleic Acids Res. 2016, Jul 27. 44(13). 6318-34. doi: 10.1093/nar/gkw565.
- Katahira J., Yoneda Y. Nucleocytoplasmic transport of microRNAs and related small RNAs. Traffic. 2011 Nov. 12(11). 1468-74. doi: 10.1111/j.1600-0854.2011.01211.x.
- Kim K.M., Abdelmohsen K., Mustapic M. et al. RNA in extracellular vesicles. Wiley Interdiscip Rev. RNA. 2017 Jul. 8(4). doi: 10.1002/wrna.1413.
- Koppers-Lalic D., Hackenberg M., Bijnsdorp I.V. et al. Nontemplated nucleotide additions distinguish the small RNA composition in cells from exosomes. Cell. Rep. 2014, Sep 25. 8(6). 1649-1658. doi: 10.1016/j.celrep.2014.08.027.
- Kosaka N., Iguchi H., Yoshioka Y. et al. Secretory mechanisms and intercellular transfer of microRNAs in living cells. J. Biol. Chem. 2010, Jun 4. 285(23). 17442-52. doi: 10.1074/jbc.M110.107821.
- LeBleu V.S., Kalluri R. Exosomes as a Multicomponent Biomarker Platform in Cancer. Trends in Cancer. 2020. 6(9). 767-774. Doi: 10.1016/j.trecan.2020.03.007.
- Leone D.A., Rees A.J., Kain R. Dendritic cells and routing cargo into exosomes. Immunol. Cell. Biol. 2018, May 24. doi: 10.1111/imcb.12170.
- Li S., Yao J., Xie M. et al. Exosomal miRNAs in hepatocellular carcinoma development and clinical responses. J. Hematol. Oncol. 2018, Apr 11. 11(1). 54. doi: 10.1186/s13045-018-0579-3.
- Li T., Yan Y., Wang B. et al. Exosomes derived from human umbilical cord mesenchymal stem cells alleviate liver fibrosis. Stem. Cells Dev. 2013, Mar 15. 22(6). 845-54. doi: 10.1089/scd.2012.0395.
- Liu H., Lei C., He Q. et al. Nuclear functions of mammalian MicroRNAs in gene regulation, immunity and cancer. Mol. Cancer. 2018, Feb 22. 17(1). 64. doi: 10.1186/s12943-018-0765-5.
- Lu J. , Wu J., Tian J., Wang S. Role of T cell-derived exosomes in immunoregulation. Immunol. Res. 2018 Jun. 66(3). 313-322. doi: 10.1007/s12026-018-9000-0.
- Luarte A., Cisternas P., Caviedes A. et al. Astrocytes at the Hub of the Stress Response: Potential Modulation of Neurogenesis by miRNAs in Astrocyte-Derived Exosomes. Stem. Cells Int. 2017. 2017. 1719050. doi: 10.1155/2017/1719050.
- Luo Y., Na Z., Slavoff S.A. P-Bodies: Composition, Properties, and Functions. Biochemistry. 2018, May 1. 57(17). 2424-2431. doi: 10.1021/acs.biochem.7b01162.
- Maji S., Matsuda A., Yan I.K. et al. Extracellular vesicles in liver diseases. Am. J. Physiol. Gastrointest. Liver Physiol. 2017, Mar 1. 312(3). 194-200. doi: 10.1152/ajpgi.00216.2016.
- Makarova J., Turchinovich A., Shkurnikov M., Tonevitsky A. Extracellular miRNAs and Cell Communication: Problems and Prospects. Trends in Biochemical Sciences. 2021. 8(46). 640-651. doi: 10.1016/j.tibs.2021.01.007.
- Makarova J.A., Shkurnikov M.U., Wicklein D. et al. Intracellular and extracellular microRNA: An update on localization and biological role. Prog. Histochem. Cytochem. 2016 Nov. 51(3–4). 33-49. doi: 10.1016/j.proghi.2016.06.001.
- Masyuk A.I., Masyuk T.V., Larusso N.F. Exosomes in the pathogenesis, diagnostics and therapeutics of liver diseases. J. Hepatol. 2013 Sep. 59(3). 621-5. doi: 10.1016/j.jhep.2013.03.028.
- McLellan A.D. Exosome release by primary B cells. Crit. Rev. Immunol. 2009. 29(3) 203-17. PMID: 19538135.
- Meldolesi J. Exosomes and Ectosomes in Intercellular Communication. Curr. Biol. 2018, Apr 23. 28(8). 435-444. doi: 10.1016/j.cub.2018.01.059.
- Nishi K., Nishi A., Nagasawa T., Ui-Tei K. Human TNRC6A is an Argonaute-navigator protein for microRNA-mediated gene silencing in the nucleus. RNA. 2013 Jan. 19(1). 17-35. doi: 10.1261/rna.034769.112.
- Nong K., Wang W., Niu X. et al. Hepatoprotective effect of exosomes from human-induced pluripotent stem cell-derived mesenchymal stromal cells against hepatic ischemia-reperfusion injury in rats. Cytotherapy. 2016 Dec. 18(12). 1548-1559. doi: 10.1016/j.jcyt.2016.08.002.
- Paris, Z. Nuclear tRNA export in trypanosomes: A journey full of twists and turns guided by tRNA modifications. Parasitology. 2021. 148(10). 1219-1222. doi: 10.1017/S0031182021000482.
- Polanco J.C., Li C., Durisic N. et al. Exosomes taken up by neurons hijack the endosomal pathway to spread to interconnected neurons. Acta Neuropathol. Commun. 2018, Feb 15. 6(1). 10. doi: 10.1186/s40478-018-0514-4.
- Politz J.C., Hogan E.M., Pederson T. MicroRNAs with a nucleolar location. RNA. 2009 Sep. 15(9). 1705-15. doi: 10.1261/rna.1470409.
- Qiu H., Shi S., Wang S. et al. Proteomic Profiling Exosomes from Vascular Smooth Muscle Cell. Proteomics Clin. Appl. 2018, Apr 24. e1700097. doi: 10.1002/prca.201700097.
- Reyes-Gutierrez P., Ritland Politz J.C., Pederson T. A mRNA and cognate microRNAs localize in the nucleolus. Nucleus. 2014. 5(6). 636-42. doi: 10.4161/19491034.2014.990864.
- Roberts T.C. The MicroRNA Biology of the Mammalian Nucleus. Mol. Ther. Nucleic. Acids. 2014, Aug 19. 3. e188. doi: 10.1038/mtna.2014.40.
- Royo F., Moreno L., Mleczko J. et al. Hepatocyte-secreted extracellular vesicles modify blood metabolome and endothelial function by an arginase-dependent mechanism. Sci. Rep. 2017, Feb 17. 7. 42798. doi: 10.1038/srep42798.
- Royo F., Schlangen K., Palomo L. et al. Transcriptome of extracellular vesicles released by hepatocytes. PLoS One. 2013, Jul 11. 8(7). e68693. doi: 10.1371/journal.pone.0068693.
- Ruivo C.F., Adem B., Silva M., Melo S.A. The Biology of Cancer Exosomes: Insights and New Perspectives. Cancer Res. 2017, Dec 1. 77(23). 6480-6488. doi: 10.1158/0008-5472.CAN-17-0994.
- Salehi M., Sharifi M. Exosomal miRNAs as novel cancer biomarkers: Challenges and opportunities. J. Cell. Physiol. 2018, Jan 11. doi: 10.1002/jcp.26481.
- Sankaranarayanan M., Emenecker R.J., Jahnel M., Trussina I.R.E.A., Wayland M., Alberti S., Holehouse A.S., Weil T.T. The arrested state of processing bodies supports mRNA regulation in early development. bioRxiv. 2021. 03(16). 435709. doi: 10.1101/2021.03.16.435709.
- Saunderson S.C., Dunn A.C., Crocker P.R., McLellan A.D. CD169 mediates the capture of exosomes in spleen and lymph node. Blood. 2014, Jan 9. 123(2). 208-16. doi: 10.1182/blood-2013-03-489732.
- Schraivogel D., Schindler S.G., Danner J. et al. Importin-β facilitates nuclear import of human GW proteins and balances cytoplasmic gene silencing protein levels. Nucleic Acids Res. 2015, Sep 3. 43(15). 7447-61. doi: 10.1093/nar/gkv705.
- Shapouri F., Saeidi S., de Iongh R.U. et al. Tob1 is expressed in developing and adult gonads and is associated with the P-body marker, Dcp2. Cell. Tissue Res. 2016 May. 364(2). 443-51. doi: 10.1007/s00441-015-2328-z.
- Smythies L.E., Smythies J.R. Exosomes in the gut. Front. Immunol. 2014, Mar 17. 5. 104. doi: 10.3389/fimmu.2014.00104.
- Stahl P.D., Raposo G. Exosomes and extracellular vesicles: the path forward. Essays Biochem. 2018, May 15. 62(2). 119-124. doi: 10.1042/EBC20170088.
- Stalder L., Heusermann W., Sokol L. et al. The rough endoplasmatic reticulum is a central nucleation site of siRNA-mediated RNA silencing. EMBO J. 2013, Apr 17. 32(8). 1115-27. doi: 10.1038/emboj.2013.52.
- Takahashi Y., Nishikawa M., Shinotsuka H. et al. Visualization and in vivo tracking of the exosomes of murine melanoma B16-BL6 cells in mice after intravenous injection. J. Biotechnol. 2013, May 20. 165(2). 77-84. doi: 10.1016/j.jbiotec.2013.03.013.
- Tan C.Y., Lai R.C., Wong W. et al. Mesenchymal stem cell-derived exosomes promote hepatic regeneration in drug-induced liver injury models. Stem. Cell. Res. Ther. 2014, Jun 10. 5(3). 76. doi: 10.1186/scrt465.
- Tkach M., Théry C. Communication by Extracellular Vesicles: Where We Are and Where We Need to Go. Cell. 2016, Mar 10. 164(6). 1226-1232. doi: 10.1016/j.cell.2016.01.043.
- Tucci M., Mannavola F., Passarelli A. et al. Exosomes in melanoma: a role in tumor progression, metastasis and impaired immune system activity. Oncotarget. 2018, Apr 17. 9(29). 20826-20837. doi: 10.18632/oncotarget.24846.
- Valadi H., Ekström K., Bossios A. et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat. Cell. Biol. 2007 Jun. 9(6). 654-9. doi: 10.1038/ncb1596.
- Varela-Eirin M., Varela-Vazquez A., Rodríguez-Candela Mateos M. et al. Recruitment of RNA molecules by connexin RNA-binding motifs: Implication in RNA and DNA transport through microvesicles and exosomes. Biochim. Biophys. Acta. 2017 Apr. 1864(4). 728-736. doi: 10.1016/j.bbamcr.2017.02.001.
- Villarroya-Beltri C., Gutiérrez-Vázquez C., Sánchez-Cabo F. et al. Sumoylated hnRNPA2B1 controls the sorting of miRNAs into exosomes through binding to specific motifs. Nat. Commun. 2013. 4. 2980. doi: 10.1038/ncomms3980.
- Vukman K.V., Försönits A., Oszvald Á. et al. Mast cell secretome: Soluble and vesicular components. Semin. Cell. Dev. Biol. 2017 Jul. 67. 65-73. doi: 10.1016/j.semcdb.2017.02.002.
- Waldenström A., Gennebäck N., Hellman U., Ronquist G. Cardiomyocyte microvesicles contain DNA/RNA and convey biological messages to target cells. PLoS One. 2012. 7(4). e34653. doi: 10.1371/journal.pone.0034653.
- Wang S., Wang J.Q., Lv X.W. Exosomal miRNAs as biomarkers in the diagnosis of liver disease. Biomark Med. 2017 May. 11(6). 491-501. doi: 10.2217/bmm-2017-0011.
- Wei Y., Li L., Wang D. et al. Importin 8 regulates the transport of mature microRNAs into the cell nucleus. J. Biol. Chem. 2014, Apr 11. 289(15):10270-5. doi: 10.1074/jbc.C113.541417.
- Willekens F.L., Werre J.M., Kruijt J.K. et al. Liver Kupffer cells rapidly remove red blood cell-derived vesicles from the circulation by scavenger receptors. Blood. 2005, Mar 1. 105(5). 2141-5. Doi: 10.1182/blood-2004-04-1578.
- Yáñez-Mó M., Siljander P.R., Andreu Z. et al. Biological properties of extracellular vesicles and their physiological functions. J. Extracell. Vesicles. 2015, May 14. 4. 27066. doi: 10.3402/jev.v4.27066.
- Yu X., Odenthal M., Fries J.W. Exosomes as miRNA Carriers: Formation-Function-Future. Int. J. Mol. Sci. 2016, Dec 2. 17(12). pii: E2028. Doi: 10.3390/ijms17122028.
- Zhang J., Li. S., Li L. et al. Exosome and exosomal microRNA: trafficking, sorting, and function. Genomics Proteomics Bioinformatics. 2015 Feb. 13(1). 17-24. doi: 10.1016/j.gpb.2015.02.001.
- Zhang T., Tan P., Wang L. RNALocate: a resource for RNA subcellular localizations. Nucleic Acids Res. 2017, Jan 4. 45(D1). 135-138. doi: 10.1093/nar/gkw728.
- Zhang W., Jiang X., Bao J. et al. Exosomes in Pathogen Infections: A Bridge to Deliver Molecules and Link Functions. Front. Immunol. 2018, Feb 12. 9. 90. doi: 10.3389/fimmu.2018.00090.
- Zhu Q., Heon M., Zhao Z., He M. Microfluidic engineering of exosomes: editing cellular messages for precision therapeutics. Lab. Chip. 2018, Jun 12. 18(12). 1690-1703. doi: 10.1039/c8lc00246k.

/52.jpg)
/53.jpg)
/54.jpg)
/55.jpg)
/56.jpg)
/57.jpg)