Журнал «Здоровье ребенка» Том 17, №3, 2022
Вернуться к номеру
Механізми дії цитоплазматичних мікроРНК. Частина 2. МікроРНК-опосередкований посттрансляційний сайленсинг
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді розглянуто механізми дії цитоплазматичних мікроРНК, а саме мікроРНК-опосередкований посттрансляційний сайленсинг. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Вказано, що синтез протеїнів є складним процесом, у реалізації якого беруть участь численні регулятори. Відомо, що процес трансляції складається з трьох основних етапів: ініціації, елонгації поліпептидного ланцюга й термінації. Показано, що в процесі ініціації трансляції беруть участь десятки «основних» факторів і численні аксесуарні протеїни, як регулятори, так і репресори процесу. Авторами наведена кінетична модель, запропонована Christopher S. Fraser. Згідно з цією моделлю ініціація трансляції є ранжируваним процесом. Підкреслено, що надалі відбувається взаємодія рибосоми з початком кодуючої нуклеотидної послідовності мРНК. Модифікації нуклеотидів факторами елонгації в антикодон тРНК регулюють динаміку функціонування рибосоми і цим тонко налаштовують швидкість синтезу протеїну. Автори показують, що термінація трансляції індукується взаємодією декодуючої А-ділянки рибосоми з одним з трьох стоп-кодонів (UAA, UAG або UGA) мРНК. У термінації трансляції також беруть участь фактори термінації. Основні фактори, які регулюють функціональну активність мРНК, діють на кеп і полі(А)хвіст, що захищають мРНК від дії екзонуклеази.Отже, різні протеїни оточують молекулу мРНК у клітині й підтримують існування і функціональну активність мРНК. Кожен регіон мРНК взаємодіє зі специфічним спектром РНК-зв’язуючих протеїнів. Ініціація трансляції є ранжируваним процесом. Ініціація трансляції і деградація мРНК нерозривно пов’язані одна з одною. Існує поширена думка про те, що трансляція в основному контролюється в періоді ініціації. Механізм сайленсингу, що обумовлений деградацією мРНК, залежить від розміру комплементарного регіону.
The scientific review presents the mechanisms of action of cytoplasmic miRNAs, namely miRNA-mediated posttranslational silencing. To write the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. It is stated that protein synthesis is a complex process which involved many regulators. It is known that the translation process consists of three main stages: initiation, elongation of the polypeptide chain and termination. It is presented that dozens of “basic” factors and numerous accessory proteins, both regulators and repressors of the process, take part in the translation initiation. The authors provide a kinetic model proposed by Christopher S. Fraser. According to this model, translation initiation is a ranked process. It is emphasized that subsequently the ribosome interacts with the beginning of the coding nucleotide sequence of mRNA. Modifications of nucleotides by elongation factors in the anticodon of tRNA regulate the dynamics of ribosome function and, thus, fine-tune the rate of protein synthesis. The authors state that translation termination is induced by the interaction of the decoding A-region of the ribosome with one of the three stop codons (UAA, UAG or UGA) of mRNA. “Termination factors” are also involved in the termination of translation. Scientists say that the main factors that regulate the functional activity of mRNA act on the cap and poly(A)tail, which protects mRNA from exonuclease action. Thus, various proteins surround mRNA molecule in the cell and support the existence and functional activity of mRNA. Each mRNA region interacts with a specific spectrum of RNA-binding proteins. The initiation of translation is a ranked process and is inextricably linked with mRNA degradation. It is widely believed that translation is largely controlled during the initiation period. The mechanism of silencing caused by mRNA degradation depends on the size of the complementary region.
мікроРНК; ініціація; трансляція; мікроРНК-опосередкований посттрансляційний сайленсинг; огляд
microRNA; miRNA; miR; initiation; translation; microRNA-mediated posttranslational silencing; review
Вступ
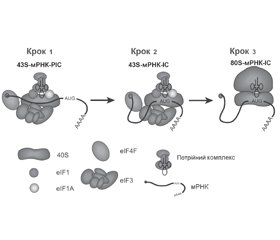
МікроРНК-опосередкований посттрансляційний сайленсинг
МікроРНК-опосередкований сайленсинг, асоційований з деградацією мРНК
Висновки
- Alkalaeva E., Mikhailova T. Reassigning stop codons via translation termination: How a few eukaryotes broke the dogma. Bioessays. 2017 Mar. 39(3). doi: 10.1002/bies.201600213.
- Andreev D.E., Dmitriev S.E., Loughran G. et al Translation control of mRNAs encoding mammalian translation initiation factors. Gene. 2018 Apr 20. 651. 174-182. doi: 10.1016/j.gene.2018.02.013.
- Bartel D.P. MicroRNAs: target recognition and regulatory functions. Cell. 2009 Jan 23. 136(2). 215-33. doi: 10.1016/j.cell.2009.01.002.
- Carbonell A. Plant ARGONAUTEs: Features, Functions, and Unknowns. Methods Mol. Biol. 2017. 1640. 1-21. doi: 10.1007/978-1-4939-7165-7_1.
- Corley M., Burns M.C., Yeo G.W. How RNA-Binding Proteins Interact with RNA: Molecules and Mechanisms. Mol Cell. 2020 Apr 2. 78(1). 9-29. doi: 10.1016/j.molcel.2020.03.011.
- Das S., Das B. eIF4G-an integrator of mRNA metabolism? FEMS Yeast Res. 2016 Nov. 16(7). pii: fow087.
- Dauden M., Jaciuk М., Müller C.W., Glatt S. Structural asymmetry in the eukaryotic Elongator complex. FEBS Lett. 2018 Feb. 592(4). 502-515. doi: 10.1002/1873-3468.12865.
- Fabian M.R., Sonenberg N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC. Nat. Struct. Mol. Biol. 2012 Jun 5. 19(6). 586-93. doi: 10.1038/nsmb.2296.
- Fraser C.S. Quantitative studies of mRNA recruitment to the eukaryotic ribosome. Biochimie. 2015 Jul. 114:58-71. doi: 10.1016/j.biochi.2015.02.017.
- Fukao A., Aoyama T., Fujiwara T. The molecular mechanism of translational control via the communication between the microRNA pathway and RNA-binding proteins. RNA Biol. 2015. 12(9). 922-6. doi: 10.1080/15476286.2015.1073436.
- Ghidini A., Cléry A., Halloy F., Allain F.H.T., Hall J. RNA-PROTACs: Degraders of RNA-Binding Proteins. Angew. Chem. Int. Ed. Engl. 2021 Feb 8. 60(6). 3163-3169. doi: 10.1002/anie.202012330.
- Hall K.B. RNA and Proteins: Mutual Respect. F1000Res. 2017 Mar 27. 6. 345. doi: 10.12688/f1000research.10572.1.
- Han S.P., Tang Y.H., Smith R. Functional diversity of the hnRNPs: past, present and perspectives. Biochem. J. 2010 Sep 15. 430(3). 379-92. doi: 10.1042/BJ20100396.
- Hao P., Yu J., Ward R., Liu Y., Hao Q., An S., Xu T. Eukaryotic translation initiation factors as promising targets in cancer therapy. Cell. Commun. Signal. 2020 Nov 4. 18(1). 175. doi: 10.1186/s12964-020-00607-9.
- Harrison A.F., Shorter J. RNA-binding proteins with prion-like domains in health and disease. Biochem. J. 2017 Apr 7. 474(8). 1417-1438. doi: 10.1042/BCJ20160499.
- Hinnebusch A.G. Molecular mechanism of scanning and start codon selection in eukaryotes. Microbiol. Mol. Biol. Rev. 2011 Sep. 75(3). 434-67, first page of table of contents. doi: 10.1128/MMBR.00008-11.
- Hinnebusch A.G. Structural Insights into the Mechanism of Scanning and Start Codon Recognition in Eukaryotic Translation Initiation. Trends Biochem. Sci. 2017 Aug. 42(8). 589-611. doi: 10.1016/j.tibs.2017.03.004.
- Hinnebusch A.G. The scanning mechanism of eukaryotic translation initiation. Annu. Rev. Biochem. 2014. 83. 779-812. doi: 10.1146/annurev-biochem-060713-035802.
- Hong S. RNA Binding Protein as an Emerging Therapeutic Target for Cancer Prevention and Treatment. J. Cancer Prev. 2017 Dec. 22(4). 203-210. doi: 10.15430/JCP.2017.22.4.203.
- Huntzinger E., Izaurralde E. Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet. 2011 Feb. 12(2). 99-110. doi: 10.1038/nrg2936.
- Ipsaro J.J., Joshua-Tor L. From guide to target: molecular insights into eukaryotic RNA-interference machinery. Nat. Struct. Mol. Biol. 2015 Jan. 22(1). 20-8. doi: 10.1038/nsmb.2931.
- Jones C.I., Zabolotskaya M.V., Newbury S.F. The 5’→3’ exoribonuclease XRN1/Pacman and its functions in cellular processes and development. Wiley Interdiscip. Rev. RNA. 2012 Jul-Aug. 3(4). 455-68. doi: 10.1002/wrna.1109.
- Loreni F., Mancino M., Biffo S. Translation factors and ribosomal proteins control tumor onset and progression: how? Oncogene. 2014 Apr 24. 33(17). 2145-56. doi: 10.1038/onc.2013.153.
- Merrick W.C., Pavitt G.D. Protein Synthesis Initiation in Eukaryotic Cells. Cold Spring Harb. Perspect. Biol. 2018 Dec 3. 10(12). a033092. doi: 10.1101/cshperspect.a033092.
- Mitchell S.F., Walker S.E., Algire M.A. et al The 5’-7-methylguanosine cap on eukaryotic mRNAs serves both to stimulate canonical translation initiation and to block an alternative pathway. Mol Cell. 2010 Sep 24. 39(6). 950-62. doi: 10.1016/j.molcel.2010.08.021.
- Nagarajan V.K., Jones C.I., Newbury S.F., Green P.J. XRN 5’→3’ exoribonucleases: structure, mechanisms and functions. Biochim. Biophys. Acta. 2013 Jun-Jul. 1829(6-7). 590-603. doi: 10.1016/j.bbagrm.2013.03.005.
- Pashler A.L., Towler B.P., Jones C.I., Newbury S.F. The roles of the exoribonucleases DIS3L2 and XRN1 in human disease. Biochem. Soc. Trans. 2016 Oct 15. 44(5). 1377-1384.
- Prabhakar A., Choi J., Wang J. et al Dynamic basis of fidelity and speed in translation: Coordinated multistep mechanisms of elongation and termination. Protein Sci. 2017 Jul. 26(7). 1352-1362. doi: 10.1002/pro.3190.
- Rissland O.S. The organization and regulation of mRNA-protein complexes. Wiley Interdiscip. Rev. RNA. 2017 Jan. 8(1). doi: 10.1002/wrna.1369.
- Shirokikh N.E., Preiss T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdiscip. Rev. RNA. 2018 Jul. 9(4). e1473. doi: 10.1002/wrna.1473.
- Stavast C.J., Erkeland S.J. The Non-Canonical Aspects of MicroRNAs: Many Roads to Gene Regulation. Cells. 2019 Nov 19. 8(11). 1465. doi: 10.3390/cells8111465.
- Zealy R.W., Wrenn S.P., Davila S. et al. microRNA-binding proteins: specificity and function. Wiley Interdiscip. Rev. RNA. 2017 Sep. 8(5). doi: 10.1002/wrna.1414.

/52.jpg)
/53.jpg)
/53_2.jpg)
/54.jpg)