Международный эндокринологический журнал Том 21, №3, 2025
Вернуться к номеру
Епігенетичний вплив довгих некодуючих РНК на розвиток інсулінорезистентності при метаболічно асоційованій жировій хворобі печінки (частина 2)
Авторы: O.E. Abaturov, A.O. Nikulina
Dnipro State Medical University, Dnipro, Ukraine
Рубрики: Эндокринология
Разделы: Справочник специалиста
Версия для печати
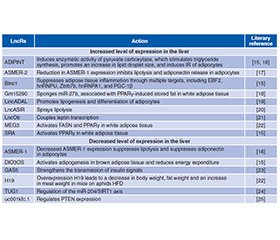
Розуміння механізмів дії довгих некодуючих РНК (long non-coding RNAs — lncR) та їхнього значення в розвитку інсулінорезистентності (ІР) у пацієнтів із метаболічно асоційованою жировою хворобою печінки дозволить модифікувати й підвищити ефективність методів діагностики і лікування метаболічних порушень. Жирова тканина є інсулінозалежною та відіграє істотну роль у метаболізмі глюкози. Збудження інсулінового рецептора (insulin receptor — INSR) адипоцитів білої жирової тканини активує транспортування глюкози, вільних жирних кислот (ВЖК), гліцерину в клітину, стимулює ліпогенез de novo й адипогенез, а також пригнічує активність механізмів ліполізу. Провідними тригерами, що викликають розвиток ІР адипоцитів, є ліпотоксичність і неспецифічне запалення жирової тканини. Зокрема, у жировій тканині хворих із метаболічно асоційованим стеатогепатитом спостерігається надекспресія мРНК TNF-α і IL-6. Медіатори запалення, як-от TNF-α та IL-1β, інгібують INSR, а TNF-α індукує фосфорилювання серинового залишку молекули IRS-1, порушуючи передачу сигналу на фосфатидилінозитол-4,5-бісфосфат-3-кіназу. Ожиріння, у свою чергу, активує мітохондріальну ВЖК-стимульовану аденіннуклеотидну транслоказу-2, що призводить до гіпоксії адипоцитів і стимулює індукований гіпоксією фактор 1α. Активація останнього викликає пригнічення поглинання глюкози адипоцитами, посилює процес гліколізу за рахунок впливу на численні ферменти, що беруть участь у метаболізмі глюкози, також індукуючи дисфункцію і запалення жирової тканини. Автори наголошують, що інсулінорезистентна жирова тканина характеризується низьким рівнем інфлюксу глюкози в адипоцити, високим рівнем вивільнення ВЖК після інсулінової стимуляції, що загалом призводить до гіперглікемії та гіперліпідемії. У патогенезі ІР білої жирової тканини беруть участь наступні lncR: ADIPINT, ASMER, Blnc1, DIO3OS, GAS5, Gm15290, H19, LncOb, MEG3, SRA та інші. Довгі некодуючі РНК, що беруть участь у патогенезі ІР м’язової тканини: IRLnc, H19, NONMMUT044897.
Understanding the mechanisms of action of long non-coding RNAs (lncR) and their significance in the development of insulin resistance (IR) in patients with metabolically associated fatty liver disease will allow modifying and increasing the effectiveness of methods for diagnosing and treating metabolic disorders. Adipose tissue is insulin-dependent and plays a significant role in glucose metabolism. The stimulation of the insulin receptor (INSR) of white adipose tissue adipocytes activates the transport of glucose, free fatty acids (FFA), and glycerol into the cell, stimulates lipogenesis de novo and adipogenesis, and also inhibits the activity of lipolysis mechanisms. The leading triggers that cause the development of IR in adipocytes are lipotoxicity and low-grade inflammation of adipose tissue. In particular, in the adipose tissue of patients with metabolically associated steatohepatitis, overexpression of TNF-α and IL-6 mRNA is observed. Inflammatory mediators such as TNF-α and IL-1β inhibit INSR, and TNF-α induces phosphorylation of the serine residue of the IRS-1 molecule, disrupting signal transmission to phosphatidylinositol-4,5-bisphosphate-3-kinase. Obesity activates mitochondrial FFA-stimulated adenine nucleotide translocase 2, which leads to hypoxia of adipocytes and stimulates hypoxia-induced factor-1α. Activation of the latter causes inhibition of glucose uptake by adipocytes, enhances the process of glycolysis by affecting numerous enzymes involved in glucose metabolism, also inducing dysfunction and inflammation of adipose tissue. The authors emphasize that insulin-resistant adipose tissue is characterized by a low level of glucose influx into adipocytes, a high level of FFA release after insulin stimulation, which generally leads to hyperglycemia and hyperlipidemia. The following lncRs are involved in the pathogenesis of IR of white adipose tissue: ADIPINT, ASMER, Blnc1, DIO3OS, GAS5, Gm15290, H19, LncOb, MEG3, SRA. Long non-coding RNAs involved in the pathogenesis of IR of muscle tissue are IRLnc, H19, NONMMUT044897.
ожиріння; інсулінорезистентність; метаболічно асоційована жирова хвороба печінки; довгі некодуючі РНК
obesity; insulin resistance; metabolically associated fatty liver disease; long non-coding RNAs
Для ознакомления с полным содержанием статьи необходимо оформить подписку на журнал.
- Gastaldelli A, Gaggini M, DeFronzo RA. Role of adipose tissue insulin resistance in the natural history of type 2 diabetes: results from the San Antonio metabolism study. Diabetes. 2017 Apr;66(4):815-822. doi: 10.2337/db16-1167.
- Petersen MC, Shulman GI. Mechanisms of insulin action and insulin resistance. Physiol Rev. 2018 Oct 1;98(4):2133-2223. doi: 10.1152/physrev.00063.2017.
- Abaturov A, Nikulina A. Predicting metabolically unhealthy obesity in children. Horm Res Paediatr. 2024;97(suppl 3):495. doi: 10.1159/000541189.
- Czech MP. Mechanisms of insulin resistance related to white, beige, and brown adipocytes. Mol Metab. 2020 Apr;34:27-42. doi: 10.1016/j.molmet.2019.12.014.
- Tkach S, Pankiv V, Dorofeev A. Relationship between serum 25-hydroxyvitamin D and non-alcoholic fatty liver disease. Int J Endocrinol (Ukraine). 2023;19(3):194-199. doi: 10.22141/2224-0721.19.3.2023.1271.
- Yang A, Mottillo EP. Adipocyte lipolysis: from molecular mecha–nisms of regulation to disease and therapeutics. Biochem J. 2020 Mar 13;477(5):985-1008. doi: 10.1042/BCJ20190468.
- Norton L, Shannon C, Gastaldelli A, et al. Insulin: the master regulator of glucose metabolism. Metabolism. 2022 Apr;129:155142. doi: 10.1016/j.metabol.2022.155142.
- Niu T, Zhu J, Dong L, et al. Inorganic pyrophosphatase 1 activates the phosphatidylinositol 3-kinase/Akt signaling to promote tumorigenicity and stemness properties in colorectal cancer. Cell Signal. 2023 Aug;108:110693. doi: 10.1016/j.cellsig.2023.110693.
- Kojta I, Chacińska M, Błachnio-Zabielska A. Obesity, bioactive lipids, and adipose tissue inflammation in insulin resistance. Nutrients. 2020 May 3;12(5):1305. doi: 10.3390/nu12051305.
- Ahmed B, Sultana R, Greene MW. Adipose tissue and insulin resistance in obesе. Biomed Pharmacother. 2021 May;137:111315. doi: 10.1016/j.biopha.2021.111315.
- Sakurai Y, Kubota N, Yamauchi T, et al. Role of insulin resistance in MAFLD. Int J Mol Sci. 2021 Apr 16;22(8):4156. doi: 10.3390/ijms22084156.
- Hai Y, Ren K, Zhang Y, et al. HIF-1α serves as a co-linker between AD and T2DM. Biomed Pharmacother. 2024 Feb;171:116158. doi: 10.1016/j.biopha.2024.116158.
- Fazakerley DJ, Krycer JR, Kearney AL, et al. Muscle and adipose tissue insulin resistance: malady without mechanism? J Lipid Res. 2019 Oct;60(10):1720-1732. doi: 10.1194/jlr.R087510.
- An T, Zhang J, Lv B, et al. Salvianolic acid B plays an anti-obesity role in high-fat diet-induced obese mice by regulating the expression of mRNA, circRNA, and lncRNA. PeerJ. 2019 Feb 28;7:e6506. doi: 10.7717/peerj.6506.
- Yang W, Lyu Y, Xiang R, et al. Long non-coding RNAs in the pathogenesis of insulin resistance. Int J Mol Sci. 2022 Dec 16;23(24):16054. doi: 10.3390/ijms232416054.
- Kerr AG, Wang Z, Wang N, et al. The long noncoding RNA ADIPINT regulates human adipocyte metabolism via pyruvate carbo–xylase. Nat Commun. 2022 May 26;13(1):2958. doi: 10.1038/s41467-022-30620-0.
- Gao H, Kerr A, Jiao H, et al. Long non-coding RNAs associa–ted with metabolic traits in human white adipose tissue. EBioMedicine. 2018 Apr;30:248-260. doi: 10.1016/j.ebiom.2018.03.010.
- Liu W, Ma C, Yang B, et al. LncRNA Gm15290 sponges miR-27b to promote PPARγ-induced fat deposition and contribute to body weight gain in mice. Biochem Biophys Res Commun. 2017 Nov 25;493(3):1168-1175. doi: 10.1016/j.bbrc.2017.09.114.
- Zhang X, Xue C, Lin J, et al. Interrogation of nonconserved human adipose lincRNAs identifies a regulatory role of linc-ADAL in adipocyte metabolism. Sci Transl Med. 2018 Jun 20;10(446):eaar5987. doi: 10.1126/scitranslmed.aar5987.
- Degirmenci U, Li J, Lim YC, et al. Silencing an insulin-induced lncRNA, LncASIR, impairs the transcriptional response to insulin signalling in adipocytes. Sci Rep. 2019 Apr 4;9(1):5608. doi: 10.1038/s41598-019-42162-5.
- Dallner OS, Marinis JM, Lu YH, et al. Dysregulation of a long noncoding RNA reduces leptin leading to a leptin-responsive form of obesity. Nat Med. 2019 Mar;25(3):507-516. doi: 10.1038/s41591-019-0370-1.
- Daneshmoghadam J, Omidifar A, Akbari Dilmaghani N, et al. The gene expression of long non-coding RNAs (lncRNAs): MEG3 and H19 in adipose tissues from obese women and its association with insulin resistance and obesity indices. J Clin Lab Anal. 2021 May;35(5):e23741. doi: 10.1002/jcla.23741.
- Shi Y, Parag S, Patel R, et al. Stabilization of lncRNA GAS5 by a small molecule and its implications in diabetic adipocytes. Cell Chem Biol. 2019 Mar 21;26(3):319-330.e6. doi: 10.1016/j.chem–biol.2018.11.012.
- Zhang Y, Gu M, Ma Y, et al. LncRNA TUG1 reduces inflammation and enhances insulin sensitivity in white adipose tissue by regulating miR-204/SIRT1 axis in obesity mice. Mol Cell Biochem. 2020 Dec;475(1–2):171-183. doi: 10.1007/s11010-020-03869-6.
- Yang L, Wang X, Guo H, et al. Whole transcriptome analysis of obese adipose tissue suggests u001kfc.1 as a potential regulator to glucose homeostasis. Front Genet. 2019 Nov 21;10:1133. doi: 10.3389/fgene.2019.01133.
- Li S, Mi L, Yu L, et al. Zbtb7b engages the long noncoding RNA Blnc1 to drive brown and beige fat development and thermoge–nesis. Proc Natl Acad Sci USA. 2017 Aug 22;114(34):E7111-E7120. doi: 10.1073/pnas.1703494114.
- Zhao XY, Li S, DelProposto JL, et al. The long noncoding RNA Blnc1 orchestrates homeostatic adipose tissue remodeling to preserve metabolic health. Mol Metab. 2018 Aug;14:60-70. doi: 10.1016/j.molmet.2018.06.005.
- Zhang R, Ma H, Gao Y, et al. Th-POK regulates mammary gland lactation through mTOR-SREBP pathway. PLoS Genet. 2018 Feb 8;14(2):e1007211. doi: 10.1371/journal.pgen.1007211.
- Cheng ZY, He TT, Gao XM, et al. ZBTB transcription factors: key regulators of the development, differentiation and effector function of T cells. Front Immunol. 2021 Jul 19;12:713294. doi: 10.3389/fimmu.2021.713294.
- Karbiener M, Fischer C, Nowitsch S, et al. MicroRNA miR-27b impairs human adipocyte differentiation and targets PPARgamma. Biochem Biophys Res Commun. 2009 Dec 11;390(2):247-251. doi: 10.1016/j.bbrc.2009.09.098.
- Srivastava A, Shankar K, Beg M, et al. Chronic hyperinsulinemia induced miR-27b is linked to adipocyte insulin resistance by targeting insulin receptor. J Mol Med (Berl). 2018 Apr;96(3–4):315-331. doi: 10.1007/s00109-018-1623-z.
- Zhou Y, Yan J, Huang H, et al. The m6A reader IGF2BP2 regulates glycolytic metabolism and mediates histone lactylation to enhance hepatic stellate cell activation and liver fibrosis. Cell Death Dis. 2024 Mar 5;15(3):189. doi: 10.1038/s41419-024-06509-9.
- Dai N, Zhao L, Wrighting D, et al. IGF2BP2/IMP2-deficient mice resist obesity through enhanced translation of Ucp1 mRNA and other mRNAs encoding mitochondrial proteins. Cell Metab. 2015 Apr 7;21(4):609-621. doi: 10.1016/j.cmet.2015.03.006.
- Wang J, Chen L, Qiang P. The role of IGF2BP2, an m6A reader gene, in human metabolic diseases and cancers. Cancer Cell Int. 2021 Feb 10;21(1):99. doi: 10.1186/s12935-021-01799-x.
- Kilpeläinen TO, Carli JF, Skowronski AA, et al. Genome-wide meta-analysis uncovers novel loci influencing circulating leptin levels. Nat Commun. 2016;7:10494. doi: 10.1038/ncomms10494.
- Abaturov A, Nikulina A. Obesity in children with leptin receptor gene polymorphisms. Acta Medica (Hradec Králové). 2021;64(3):158-164. doi: 10.14712/18059694.2021.27.
- German JP, Wisse BE, Thaler JP, et al. Leptin deficiency causes insulin resistance induced by uncontrolled diabetes. Diabetes. 2010;59(7):1626-1634. doi: 10.2337/db09-1918.
- Zhao S, Li N, Zhu Y, et al. Partial leptin deficiency confers resistance to diet-induced obesity in mice. Mol Metab. 2020 Jul;37:100995. doi: 10.1016/j.molmet.2020.100995.
- Umano GR, Cirillo G, Sanchez G, et al. The lncOb rs10487505 polymorphism impairs insulin sensitivity and glucose tole–rance in children and adolescents with obesity. Obesity (Silver Spring). 2023 Sep;31(9):2359-2364. doi: 10.1002/oby.23835.
- Manco M, Crudele A, Mosca A, et al. LncOb rs10487505 variant is associated with leptin levels in pediatric non-alcoholic fatty liver disease. Pediatr Res. 2022 Dec;92(6):1737-1743. doi: 10.1038/s41390-022-02032-9.
- Cheng X, Shihabudeen Haider Ali MS, Moran M, et al. Long non-coding RNA Meg3 deficiency impairs glucose homeostasis and insulin signaling by inducing cellular senescence of hepatic endothelium in obesity. Redox Biol. 2021 Apr;40:101863. doi: 10.1016/j.redox.2021.101863.
- Sheng L, Ye L, Zhang D, et al. New insights into the long non-coding RNA SRA: physiological functions and mechanisms of action. Front Med (Lausanne). 2018 Sep 6;5:244. doi: 10.3389/fmed.2018.00244.
- Liu S, Sheng L, Miao H, et al. SRA gene knockout protects against diet-induced obesity and improves glucose tolerance. J Biol Chem. 2014 May 9;289(19):13000-13009. doi: 10.1074/jbc.M114.564658.
- Chen G, Yu D, Nian X, et al. LncRNA SRA promotes hepatic steatosis through repressing the expression of adipose triglyceride lipase (ATGL). Sci Rep. 2016 Oct 19;6:35531. doi: 10.1038/srep35531.
- Kochumon S, Arefanian H, Sindhu S, et al. Expression of steroid receptor RNA activator 1 (SRA1) in the adipose tissue is associated with TLRs and IRFs in diabesity. Cells. 2022 Dec 11;11(24):4007. doi: 10.3390/cells11244007.
- Yang W, Li D, Wang G, et al. Expression and imprin–ting of DIO3 and DIO3OS genes in Holstein cattle. J Genet. 2017 Jun;96(2):333-339. doi: 10.1007/s12041-017-0780-0.
- Chen YT, Yang QY, Hu Y, et al. Imprinted lncRNA Dio3os preprograms intergenerational brown fat development and obesity resistance. Nat Commun. 2021 Nov 25;12(1):6845. doi: 10.1038/s41467-021-27171-1.
- Wang CY, Yu TY, Shih SR, et al. Low total and free triiodothyronine levels are associated with insulin resistance in non-diabetic individuals. Sci Rep. 2018 Jul 16;8(1):10685. doi: 10.1038/s41598-018-29087-1.
- Spira D, Buchmann N, Dörr M, et al. Association of thyroid function with insulin resistance: data from two population-based studies. Eur Thyroid J. 2022 Feb 28;11(2):e210063. doi: 10.1530/ETJ-21-0063.
- Russo SC, Salas-Lucia F, Bianco AC. Deiodinases and the metabolic code for thyroid hormone action. Endocrinology. 2021 Aug 1;162(8):bqab059. doi: 10.1210/endocr/bqab059.
- Liu C, Yang Z, Wu J, et al. Long noncoding RNA H19 interacts with polypyrimidine tract-binding protein 1 to reprogram hepa–tic lipid homeostasis. Hepatology. 2018 May;67(5):1768-1783. doi: 10.1002/hep.29654.
- Huang Y, Zheng Y, Jin C, et al. Long non-coding RNA H19 inhibits adipocyte differentiation of bone marrow mesenchymal stem cells through epigenetic modulation of histone deacetylases. Sci Rep. 2016 Jun 28;6:28897. doi: 10.1038/srep28897.
- Greene NP, Brown JL, Rosa-Caldwell ME, et al. Skeletal muscle insulin resistance as a precursor to diabetes: beyond glucoregulation. Curr Diabetes Rev. 2018;14(2):113-128. doi: 10.2174/1573399813666161122123636.
- Sylow L, Tokarz VL, Richter EA, et al. The many actions of insulin in skeletal muscle, the paramount tissue determining glycemia. Cell Metab. 2021 Apr 6;33(4):758-780. doi: 10.1016/j.cmet.2021.03.020.
- Da Silva Rosa SC, Nayak N, Caymo AM, et al. Mechanisms of muscle insulin resistance and the cross-talk with liver and adipose tissue. Physiol Rep. 2020 Oct;8(19):e14607. doi: 10.14814/phy2.14607.
- Liu Z, Zhang Z, Song G, et al. Resveratrol alleviates skeletal muscle insulin resistance by downregulating long noncoding RNA. Int J Endocrinol. 2022 Jan 19;2022:2539519. doi: 10.1155/2022/2539519.
- Wang L, Zhou ZY, Zhang T, et al. IRLnc: a novel functio–nal noncoding RNA contributes to intramuscular fat deposition. BMC Genomics. 2021 Feb 1;22(1):95. doi: 10.1186/s12864-020-07349-5.
- Gao Y, Wu F, Zhou J, et al. The H19/let-7 double-negative feedback loop contributes to glucose metabolism in muscle cells. Nucleic Acids Res. 2014 Dec 16;42(22):13799-13811. doi: 10.1093/nar/gku1160.
- Borg ML, Massart J, De Castro Barbosa T, et al. Modified UCN2 peptide treatment improves skeletal muscle mass and function in mouse models of obesity-induced insulin resistance. J Cache–xia Sarcopenia Muscle. 2021 Oct;12(5):1232-1248. doi: 10.1002/jcsm.12746.
- Liu Q, Zhu X, Xu L, et al. 6-mercaptopurine augments glucose transport activity in skeletal muscle cells in part via a mechanism dependent upon orphan nuclear receptor NR4A3. Am J Physiol Endocrinol Metab. 2013 Nov 1;305(9):E1081-1092. doi: 10.1152/ajpendo.00169.2013.
- Walton RG, Zhu X, Tian L, et al. AP2-NR4A3 transgenic mice display reduced serum epinephrine because of increased catecho–lamine catabolism in adipose tissue. Am J Physiol Endocrinol Metab. 2016 Jul 1;311(1):E69-81. doi: 10.1152/ajpendo.00330.2015.
- Fu Y, Luo L, Luo N, et al. NR4A orphan nuclear receptors modulate insulin action and the glucose transport system: potential role in insulin resistance. J Biol Chem. 2007 Oct 26;282(43):31525-31533. doi: 10.1074/jbc.M701132200.
- Pearen MA, Goode JM, Fitzsimmons RL, et al. Transge–nic muscle-specific Nor-1 expression regulates multiple pathways that affect adiposity, metabolism, and endurance. Mol Endocrinol. 2013 Nov;27(11):1897-1917. doi: 10.1210/me.2013-1205.
- Martínez-González J, Cañes L, Alonso J, et al. NR4A3: A key nuclear receptor in vascular biology, cardiovascular remodeling, and beyond. Int J Mol Sci. 2021 Oct 21;22(21):11371. doi: 10.3390/ijms222111371.
- Lee AD, Hansen PA, Schluter J, et al. Effects of epinephrine on insulin-stimulated glucose uptake and GLUT-4 phosphorylation in muscle. Am J Physiol. 1997 Sep;273(3 Pt 1):C1082-1087. doi: 10.1152/ajpcell.1997.273.3.C1082.
- Galic S, Sachithanandan N, Kay TW, et al. Suppressor of cytokine signalling (SOCS) proteins as guardians of inflammatory responses critical for regulating insulin sensitivity. Biochem J. 2014 Jul 15;461(2):177-188. doi: 10.1042/BJ20140143.
- Du Z, Uversky VN. A comprehensive survey of the roles of highly disordered proteins in type 2 diabetes. Int J Mol Sci. 2017 Sep 21;18(10):2010. doi: 10.3390/ijms18102010.
- Tkach S, Pankiv V, Krushinska Z. Features of type 2 diabetes combined with metabolic dysfunction-associated fatty liver di–sease under conditions of chronic stress. Int J Endocrinol (Ukraine). 2024;20(1):18-24. doi: 10.22141/2224-0721.20.1.2024.1353.
- Geng T, Liu Y, Xu Y, et al. H19 lncRNA promotes skeletal muscle insulin sensitivity in part by targeting AMPK. Diabetes. 2018 Nov;67(11):2183-2198. doi: 10.2337/db18-0370.
- Steinberg GR, Hardie DG. New insights into activation and function of the AMPK. Nat Rev Mol Cell Biol. 2023 Apr;24(4):255-272. doi: 10.1038/s41580-022-00547-x.
- Besse-Patin A, Jeromson S, Levesque-Damphousse P, et al. PGC1A regulates the IRS1:IRS2 ratio during fasting to influence hepatic metabolism downstream of insulin. Proc Natl Acad Sci USA. 2019 Mar 5;116(10):4285-4290. doi: 10.1073/pnas.1815150116.
- Dey BK, Pfeifer K, Dutta A. The H19 long noncoding RNA gives rise to microRNAs miR-675-3p and miR-675-5p to promote skeletal muscle differentiation and regeneration. Genes Dev. 2014 Mar 1;28(5):491-501. doi: 10.1101/gad.234419.113.
- Lewis A, Lee JY, Donaldson AV, et al. Increased expression of H19/miR-675 is associated with a low fat-free mass index in patients with COPD. J Cachexia Sarcopenia Muscle. 2016 Jun;7(3):330-44. doi: 10.1002/jcsm.12078.